實驗室開發工具
網頁工具
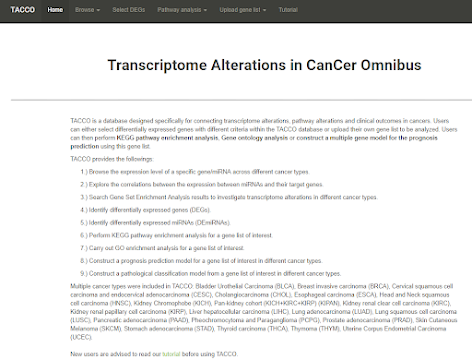
DoSurvive
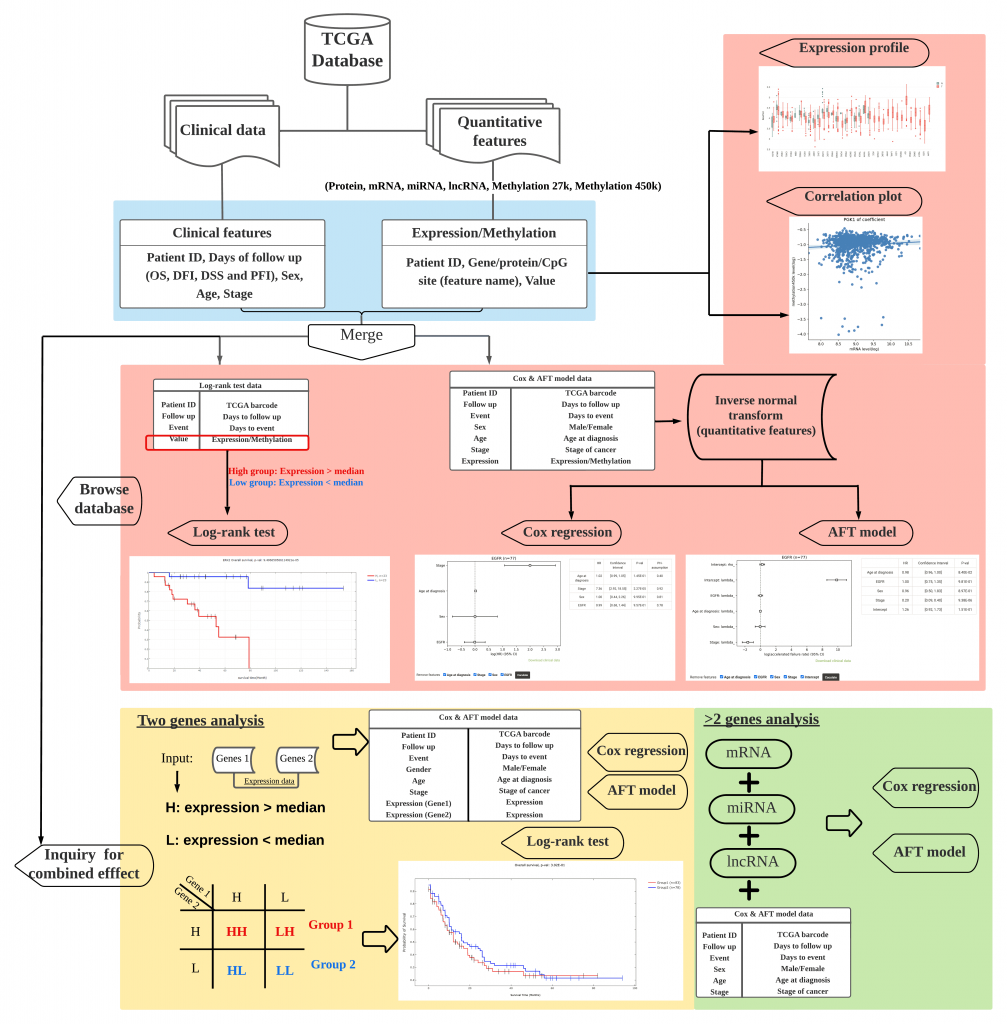
DoSurvive 提供包括mRNA, miRNA, lncRNA, protein and Methylation與癌症病人存活的單變量關聯分析結果,使用者也可線上使用TCGA資料即時進行多變量存活分析。此外,DoSurvive提供了包括Log-rank test, Cox regression和AFT三種線上存活分析服務,讓使用者可以分析自己的資料(在資料違反Proportional Hazard時可使用Log-rank test或以AFT進行多變量分析)。
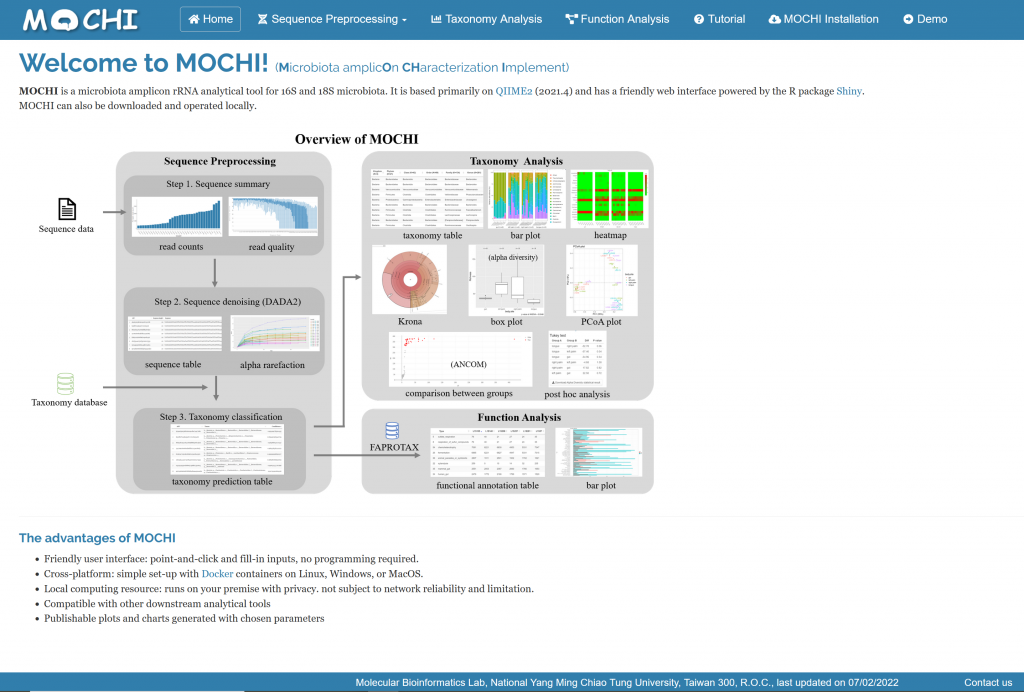
MOCHI
MOCHI為針對微生物群(microbiota)中的16S或18S rRNA擴增子(microbiota amplicon)開發之分析工具
- 使用者可從原始定序資料或taxa table開始分析
- 提供圖形化的操作介面,進行資料處理、統計分析以及繪製圖表
- 提供Docker containers的本地(local)版本,可以在Linux、Windows及MacOS的作業系統上使用
- 使用本地版的MOCHI可使用本地計算資源,確保資料安全性,並不受網路速度限制。
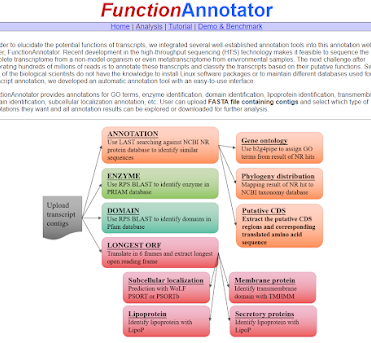
FUNCTIONANNAOTATOR
本網頁工具結合多項預測工具,提供對非模式物種的轉錄體定序後組出的contig進行功能註解,預測分析項目包括膜蛋白、酵素蛋白、細胞內位置 (subcellular localization)、GO (Gene ontology)功能註解等。
CHIPSEEK
協助分析ChIP (Chromatin Immunoprecipitation) 資料的生資網頁工具,提供peak註解、binding motif預測、peak區域比對、peak篩選等功能。
資料庫

CHLOROMITOCU
針對葉綠體、粒線體的密碼子使用頻率專門建構之資料庫,提供資料查詢外,使用者亦可自行上傳序列進行分析,並提供密碼子使用頻率之比對。
VIP DB
這是一個專門分析、比對病毒中蛋白質結構域(protein domain)分布情況的資料庫,使用者可以從此資料庫中找到某特定結構域在病毒、植物、動物中使用的情況,或者某兩個結構域是否曾一同出現在相同的病毒基因體內。